Il settore della ricerca farmaceutica è oggi in crisi e lo sviluppo di un nuove terapie richiede in media 13 anni con costi vicini al miliardo di euro. Spesso, per carenza di informazioni nelle fasi iniziali dello studio, si rendono necessari in corso d’opera cambi di strategia nel processo di progettazione farmaceutica, con sforzi e costi lievitati.
Lo scenario in cui si colloca il progetto è caratterizzato dalla presenza di un’enorme quantità di dati (big data) in campo biomedico, che risultano però di difficile consultazione e manipolazione. Le difficoltà sono collegate alla frammentarietà dei dati fruibili che non consentono al ricercatore di avere una visione d’insieme delle problematiche legate alla patologia di interesse nonché alla scarsa interoperabilità di essi. I dati oggi disponibili si presentano infatti in una varietà di formati diversi, spesso non strutturati (contenenti dati testuali e altri dati non numerici) e sono ricchi di valori mancanti. Sono dati disordinati, a volte contengono incongruenze e rumore.
Questi attributi obbligano i ricercatori a trovare modi creativi per continuare a sostenere la ricerca e lo sviluppo di terapie efficaci. Di conseguenza, c’è un’esigenza emergente per gli analisti e i produttori di big data di strumenti e tecniche informatiche che siano in grado di gestire dati rumorosi e di presentare risultati ai soggetti interessati in modo semplice e facile da interpretare. Risulta pertanto necessario, ai fini della ricerca biomedica, un sistema integrato di informazioni che, attraverso nuovi algoritmi combinati con database opportunamente federati, faciliti i processi di screening.
Il progetto si propone la realizzazione di un sistema informatizzato in campo oncologico che consenta una rapida ed efficace aggregazione e successiva analisi di data set eterogenei, focalizzata allo studio delle interazioni tra diverse molecole biologiche, che risultano alterate nelle patologie tumorali al fine di creare un disegno razionale nella progettazione di nuove terapie. I data set che si intende aggregare appartengono a due vasti ambiti di ricerca, ovvero la chimica medicinale e la trascrittomica/proteomica. Ad oggi non esiste uno strumento informatico che integri le informazioni disponibili relative a tali discipline, nonostante gli argomenti siano strettamente interlacciati. Un sistema di raccolta e analisi dati così costituito, faciliterebbe la razionale progettazione di terapie altamente specializzate e personalizzate.
L’obiettivo principale del progetto è pertanto la creazione di un sistema esperto per la valorizzazione e l’integrazione di dati già in possesso dei partner, di dati provenienti da database di pubblico dominio, e di dati che verranno via via prodotti durante le fasi di progetto attraverso algoritmi di predizione proprietari.
Fruitori del sistema sviluppato saranno operatori del settore, ricercatori della comunità medico-scientifica, aziende interessate, contributor del settore che hanno fornito i dati raccolti, enti terzi interessati ai dati elaborati, ai risultati e indicazioni raggiunte, etc. I fruitori potranno accedere al repository (dati, risultati ottenuti integrando e processando le informazioni raccolte, suggerimenti per ulteriori raccolte di dati finalizzate ad ottimizzazione delle soluzioni già individuate o all’individuazione di nuove soluzioni), da un punto qualsiasi della rete Internet attraverso moderne webGUI, attraverso le app per smartphone e tablet predisposte nell’ambito del progetto, attraverso interfacce aperte che aprono l’accesso ad altri sistemi.
Lo studio delle patologie complesse, quali sono quelle tumorali, necessita un approccio di analisi dati integrato come quello previsto dal sistema informatizzato proposto. Un gran numero di geni con diverse funzioni fisiologiche, possono essere coinvolti nello sviluppo della patologia tumorale nell’uomo. Più di 500 geni sono stati identificati come fortemente implicati nel processo di trasformazione delle cellule normali in cellule tumorali.
L’espressione di questi geni, nelle cellule sane, contribuisce alla normale crescita, alla sopravvivenza e all’espletamento delle normali funzioni biochimiche necessarie alla sopravvivenza cellulare. Una variazione nel normale processo di espressione di questi geni, inclusa l’eccessiva espressione, la perdita di espressione o l’espressione di un prodotto difettoso, dovuto ad esempio ad una mutazione puntiforme nella sequenza, può essere alla base di una errata regolazione del processo fisiologico di duplicazione cellulare, fino ad arrivare ad una crescita incontrollata del tessuto, tipico fenomeno della patologia tumorale. L’alterazione dell’espressione genica può essere causata da ri-arrangiamenti cromosomici strutturali, amplificazioni di specifiche sequenze geniche, silenziamento della trascrizione attraverso metilazione e mutazioni, ad es. mutazioni puntiformi e piccoli inserti o delezioni, che portano alla perdita o al guadagno di funzione della proteina corrispondente. Attualmente, oltre l’1% di tutti i geni umani sono implicati attraverso una mutazione nel cancro. Di questi circa il 90% ha mutazioni somatiche nel cancro, 20% mutazioni germinali che predispongono al cancro e il 10% mostra sia mutazioni somatiche che germinali. È chiaro che siamo lontani dal fatto di avere un catalogo completo di geni del cancro e risultati provenienti dagli sforzi per identificare le alterazioni genetiche implicate nel cancro sono continuamente pubblicate. In un recente studio del Broad Institute (Lawrence MS et al., 2014), viene presentato un elenco di 260 geni tumorali notevolmente mutanti su 21 tipi di cancro. In un altro studio inteso a individuare i geni del cancro (Tamborero D et al., 2013), viene presentato un elenco di 291 geni coinvolti in 12 tipi di cancro.
L’analisi dell’espressione di proteine usando esperimenti di immunoistochimica permette di visualizzare in vari tessuti tumorali la distribuzione e la relativa abbondanza di proteine codificate da geni deregolati nel cancro. Alcune proteine mostrano un’espressione differenziale tra diverse forme di cancro o tumori individuali nella stessa forma di cancro, mentre altre proteine mostrano un’espressione variabile all’interno di diversi compartimenti del tumore o in cellule tumorali di diversi stadi. Inoltre, le differenze nell’espressione possono essere trovate tra i tumori e le rispettive controparti tissutali non affette dalla patologia.
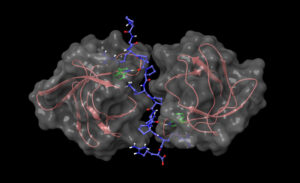
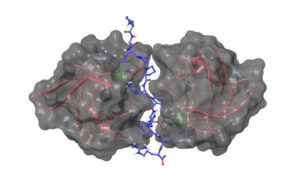
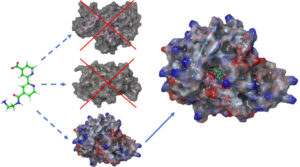
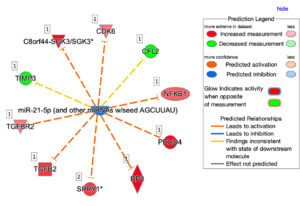
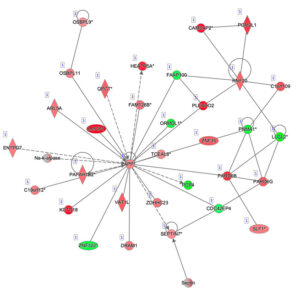
Il sistema informatizzato che si vuole realizzare sarà in particolare rivolto all’analisi delle interazioni biologiche coinvolte nell’insorgenza di un tumore, in particolare delle interazioni tra proteine, RNA e piccole molecole, valorizzando la presenza nel web di numerosi data set utilizzabili per tali analisi. L’analisi di tali interazioni, grazie alla disponibilità di tecnologie di indagine altamente sofisticate, ha sviluppato un forte carattere quantitativo su aspetti complementari:
- Rilevazione high-throughput dei livelli di espressione delle molecole biologiche (RNA, proteine) nel contesto cellulare di interesse
- Accesso a strutture tridimensionali delle molecole biologiche e in particolare delle proteine, RNA e piccole molecole
- Disponibilità di risorse computazionali e programmi di calcolo dedicati
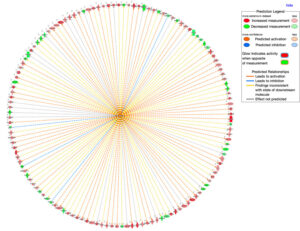
Grazie a tale disponibilità di dati, le interazioni biologiche possono essere studiate allargando l’orizzonte ad un network di interazioni quanto più ampio possibile. Qualsiasi molecola biologica, infatti, non interagisce esclusivamente con una sola controparte. È importante quindi studiare il comportamento dell’intero network, e, in questo modo, riuscire a prevedere gli effetti di una terapia su una scala più ampia della singola interazione. Obiettivo del sistema informatizzato sarà guidare il fruitore con approcci di tipo data driven e knowledge driven nella ricerca dei dati necessari per studiare il tessuto tumorale e il relativo network di interazione di interesse, dando quindi la possibilità di predire quali modifiche sul network possono essere considerate una terapia. Tale obiettivo sarà raggiunto grazie all’implementazione di una infrastruttura informatica che include le seguenti funzionalità:
- Piattaforma hardware, software, di networking, in cloud computing con funzionalità di fault, system & application management, scalabile e con capacità di ripartizione del carico di richieste se necessario;
- Data collecting dei dati significativi da qualsiasi fonte (upload di dati effettuato dai ricercatori, database esterno) con frequenze e modalità varie. I dati saranno inseriti in repository interni del sistema ICT progettati specificamente
- Sviluppo di repository interni che abilitano tutte le funzioni richieste ai dati del sistema ICT e che possono evolvere rapidamente verso modalità Open data e Big Data
- Data processing sulla base di: opportunità di aggregazione, elaborazioni di ottimizzazione, business logic, algoritmi, che saranno progettati all’interno del progetto o individuati nelle analisi preliminari
- Individuazione di eventi significativi per l’ambito progettuale: nuovi insiemi di sintomi/parametri che individuano l’insorgenza della patologia, nuove indicazioni terapeutiche, feedback sull’insorgenza di patologie o sulle indicazioni terapeutiche in essere, modifiche degli algoritmi/business logic/aggregazioni di dati, ecc.
- Disponibilità di un motore ontologico specializzato per l’accesso guidato e focalizzato ai dati del sistema ICT
- Funzioni di accesso controllato e base proxy server, secondo moderne funzionalità AAA (Authentication, Authorization, Accounting) di controllo degli accessi al sistema, alle sue funzioni e ai suoi dati
- Interfacciamento applicativo automatico verso database esterni e possibilità di
- Interfaccia easy-to-use web based responsive e app fruibili da smartphone e tablet